بسم الله الرحمن الرحيم
في المقال السابق رأينا كيف أن نجاح مشروع ترميز الجينوم البشري ساهم في فتح باب أوسع للبحث والتسآئل من أهمهما "كيف يمكننا تفسير هذه السلسلة الطويلة من الرموز وكيف يمكننا إستخلاص معلومات منها؟". فالحصول على مجموعة من A,C,G,T لايكفي لإستخراج أي أية نتائج بطريقة بديهية. لكن من المؤكد أن نهاية هذا العمل أدت بنا إلى الخروج من "عصر الجينوم" ,الذي بدأ مع إكتشاف بنية الحمض النووي من طرف واتسون وكريك سنة 1953, و الدخول إلى "عصر مابعد الجينوم" (Post-genomic era) .
كما قلنا سابقا, إعتُمِدَ في تشفير الجينوم علي التقنية المُطَوَرة من طرف فريدرك سانغر من أجل تحديد سلسلة أجزاء الحمض النووي. بإختصار, مبدأ هذه التقنية بسيط حيث يتم أخذ الجزء المراد تشفيره ثم يتم عمل عدة نسخ منه باستعمال البكتيريا, بعدها يتم وضع جزء من هذه النسخ في محلول يحتوي على كل الأزواج القاعدية لكن ال A معدل بطريقة تجعل عملية نسخ الحمض النووي تتوقف عندما يظاف هذا النكليوتيد المعدل كيمائيا مما يسمح لنا بمعرفة كل مواقع A على شريط الحمض النووي. نقوم بنفس التجربة على بقية الحروف فتمكن بذالك من معرفة كل المواقع وذالك بترسيب نسخ الحمض النووي على حسب الوزن
ولأن هذه التقنية فعالة فقط مع السلاسل الصغيرة يجب علينا تقسيم الحمض النووي إلى عدة إجزاء صغيرة والقيام بالتجربة, بطبيعة الحال تكرر التجربة عدة مرات لتفادي أية أخطاء, ثم يتم ترتيب هذه الأجزاء ويسمى هذا النوع من التشفير (أو الترميز) بـ: Short-gun sequencing .
من الأسئلة التي يمكن أن تُطرح هي: بعد تقطيع الحمض النووي إلى أجزاء كيف يمكننا جمع هذه الأجزاء وفقا لترتيبها الصحيح ؟ تخيل نفسك أمام كتاب ممزق إلى أجزاء صغيرة وطلب منك تجميعه, ولنزيد الأمور تعقيدا تخيل أن هذا الكتاب مكتوب بلغة لاتفهمها, ستبدوا أنها ضرب من المستحيل دون اللجوء إلى طريقة أوتوماتيكية تسمح لك بترتيب الكتاب بطريقة صحيحة . لهذا الغرض صممت عدة خوارزميات لتجميع السلاسل (Sequence Assembly algorithms).
نعود إلى الترميز, للقيام بهذه العملية بطريقة أوتوماتيكية تم صناعة الجيل الأول (First Generation sequencer) من المرمزات من طرف شركة Applied Biosystems والمتمثلة في جهاز DNA Analyzed 3730 و الذي يعتمد في مبدأه على إعطاء كل حرف لون محدد بعدها يقوم الجهاز بعملية نسخ الحمض النووي بستعمال هذه المركبات ثم ترتيب النسخ المتحصل عليها من الأكبر إلى الأصغر ثم قرائة الأحرف بإستعمال الليزر وإرسال المعلومات إلى الكميوتر ليتم تحليلها. يستطيع هذا النوع من الآلآت ترميز حوالي 700 جزيئ قاعدي (Base paire إختصارا bp ) في المرة الواحدة. هذه العملية و إن كانت أتوماتيكية نوعا ما إلا أنها تستلزم التدخل اليدوي من أجل تحضير الحمض النووي وعمل نسخ من السلسة المرجو تشفيرها بإستعمال البكتيريا. ونَظَرِيا يتطلب حوالي 6.3 سنة لآلة واحدة من أجل تشفير جينوم بشري, إلا أنه في مراكز البحث تستعمل عدة آلات لإختزال هذا الوقت لعدة أشهر أو أسابيع لكن بتكلفة عالية.
تمثل سنة 2004 سنة تحول رهيب في تقنيات التشفير حيث تم تطوير ألآت الجيل الثاني من المرمزات (أو المسلسِلات ) (Second Generation Sequencer) حيث خلافا لطريقة سانغر فهذه الآلآت تعتمد على طريقة ال Sythesis حيث يمكننا تصميم نكليوتيدات (يعني A,C,G,T) تصدر ضوئأ عندما تلتحم مع الحمض النووي وبهذا يمكننا في كل مرة وضع نوع واحد من النكليتوتيدات ونراقب هل يشع أم لا, وبتطبيق هذه الطريقة بطريقة متوازية على عدة سلاسل من الحمض النووي في وقت واحد يمكننا إختزال العملية من سنوات إلى أيام بإستعمال آلة واحدة.
أول آلة إعتمدت هذا المبدأ هي 454 Titanium حيث كان بإمكانها ترميز جينوم بشري في مدة 3.75 يوم ثم تلتها بعد ذالك آلات Illumina بسرعة جينوم بشري في 1.7 يوم ثم تلتها مجموعة من الآلات أكثر سرعة إلى غاية تطوير ألات الجيل الثالث التي تكمل ترميز جينوم بشري في ساعتين فقط وهناك جهود لتطوير آلات أسرع وبدقة عالية. سأحاول إنشاء الله تخصيص مقال أشرح فيه مبدأ هذه الآلات, النقطة التي أريد إيصالها هي أنه الآن لم يعد الترميز مشكلا.
هذا التطور السريع في آلات الترميز جلب معه عدة تحديات من جانب تحليل البيانات حيث تطلب الوضع تطوير عدة تقنيات إحصائية ورياضية لتصحيح المقاييس المــأخوذة من طرف الآلة و للمقارنة بين نتائج عدة تجارب وتطوير برامج وخوارزميات من أجل فهرسة هذه البيانات بطرية سريعة و ترتيبها وتحليلها ...إلخ مما أدى إلى ظهور علم المعلوماتية الحيوية و البيولوجيا الحاسوبية (Computational Biology ) وعدة تخصصات أخرى حيث أصبح من الصعب التفريق بين التخصصات فتسمى تارة المعلوماتية الحيوية وتارة البرمجية البيولوجية.
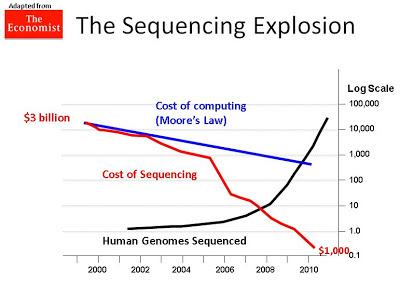
هذا التطور كان بشكل سريع جدا, حيث كان أسرع من قانون تطور سرعة الكمبيوتر المسمى بقانون مور (Moore's law) والذي يقول بـأن سرعة الكمبيوتر تتضاعف كل سنتين, مما سبب عدة تحديات حيث أن سرعة تدفق البيانات أكبر من سرعة تحليلها. لكن الخبر السار هو أن سعر تحليل الجيونوم إنخفض هو أيضا بشكل سريع مما جعل العلماء يطمحون في أن يصل سعر تشفير الجينوم إلى ألف دولار حيث سيصبع بإمكان الإنسان العادي تحليل جينومه مما يسهل إدخال هذه التقنية إلى المستشفيات وجعلها شيئا روتينيا. وبذالك ندخل في عصر الطب الشخصي (Personalized Medecine ) حيث سيصبح بإمكان الطبيب إعطاء المريض الدواء المناسب باللجرعة المناسبة بإستعمال المعلومات الموجودة في جينومه معلومات أخرى سوف نتطرق إليها في مقالات أخرى.
هذا التطور في آلات الترميز أيضا ساهم في تسريع برنامج ENCODE أو موسوعة عناصر الحمض النووي, وهو برنامج أطلق سنة 2003 بعد الإنتهاء من برنامج الجينوم البشري لمحاولة فهم ال 1% المرمزة للبروتين في الجينوم البشري وذلك لأرشفة السلاسل التي تقوم بوضائف محددة و تحديد متى تنشط هذه السلاسل وفي أية نوع من الخلايا. ظهور آلآت الجيل الثاني جعل العلماء يقررون دراسة المرور من دراسة 1% من الجينوم إلى دراسة كامل الجينوم والغوص في بحر النكليوتدات التي تملئ الفراغ بين الجينات والتي وصفت على أنها نفايات. وتمكن العلماء في مارس من سنة 2012 من إكمال المشروع وإنتاج 5 أضعاف البيانات التي خطط لإنتاجها دون أي تغيير في الميزانية. وتمت دراسة 174 نوعا مختلفا من الخلايا وجمع الكثير من البيانات. من أهم النتائج التي توصل إلها المشروع أن 80% من الجينوم نَشِطة كيميائيا , لكن ظهر إختلاف بين العلماء حول القول بأن هذه 80% تقوم بوظيفة أم فقط نشطة. لأن بعضهم يؤمن بأن الأجزاء الوظيفية يجب أن تكون محفوظة بين السلاسلات.
تم نشر نتائج هذا المشروع في ثلاثين مقالا علميا نشرت في يوم واحد في عدة مجلات علمية وهي متوفرة مجانا.
ساحاول إن شاء الله في مقلات أخرى التعريف بالتقنيات المستعملة في الترميز وأيضا التقنيات المستعملة في قياس نسبة نشاط الجينات وكيفية معالجة هذه البيانات. لكن أظن أنه قبل المرور إلى البرمجة يجب أولا مجموعة من المقالات لتوضيح بعض المبادئ في الخلية والتقنيات المستعملة لتسهيل الفهم للمهتمين بهذا المجال والذين لم يدرسوا بالضرووة علوم الأحياء.
رابط المقالة : المعلوماتية الحيوية بالعربية » مدخل إلى علم المعلوماتية الحيوية (Bioinformatics) (الجزء الثاني)