إن البحث العلمي في زمننا هذا يشهد تطورا ليس له مثيل في تاريخ البشرية حيث أن النظريات والمبادىء التي إستحدثت قبل سنتين قد أصبحت الآن في خبر كان و إستبدلت أو عدلت لتصبح أكثر فعالية. ومن نتائج هذا التطور هو إدخال تكنولجيا الحاسوب و ذالك في نظري يعود لسببين :
- الأول هو مرور العالم من زمن كان المشكل فيه الحصول على المعلومة إلى زمن يحتوي على فيض من المعلومات بحيث أن أصغر مختبر أو شركة الآن (على الأقل في العالم المتقدم) تنتج في إسبوع واحد جيغابيتات أو تيرابيتات من البيانات مما يجعل التحليل اليدوي لهذه البيانات ضربا من ضروب المستحيل ولهذا إستلزم إنشاء برامج وخوارزميات تقوم بتحليل هذه البيانات ومساعدة العالم أو مدير الشركة على فهم بيانته
- والسبب الثاني في رأيي هو سهولة التخزين و البحث و النسخ حيث يمكن تقصي المعلومات السابقة في ظروف ثواني بالإضافة إلى سهولة تحديثها و عمل عدة نسخ منها.
وعلم الأحياء لا يشذ عن غيره من العلوم في هذا المجال, حيث انه مع البدايات الأولى لظهور الحاسوب أبدى العلماء تلهفهم لإستغلال هذه التكنولوجيا. حيث أن الدكتور روبرت ليدلي (Robert Lidly ) الذي قام في سنة 1965 بنشر مقال مٌأثر في مجلة Science العلمية بعنوان أساسيات التفكير في التشحيص الطبي "Reasoning Foundations of Medical Diagnosis" حيث دعي إلى إستعمال التكنولوجيات الرقمية في مجال الطب والأحياء. وفي سنة 1984 ساهم مع العالمة مارغريت دايهوف (Margaret Oakley Dayhoff) في تأسيس قاعدة البيانات PIR والتي تحتوي على جميع سلاسل البروتين المعروفة في ذالك الوقت المدونة في "أطلس سلاسل وبنيات البروتين" المؤلف من طرف دايهوف سنة 1970.
مع تطور تكنولوجيا الإتصال و ظهور الانترنت تمكنت العديد من الجامعات و مراكز الأبحاث من الاتصال بالإنترنت مما جعل هذا النوع من قواعد البيانات متاحا للعديد من فرق البحث و أصبح لهذه قواعد البيانات دورا مهما في مختلف البحوث البيولوجية.
هذا التطور الجديد غيَّر الكثير من المعطيات في مجال الأحياء, حيث سمح التوفر السهل لهذه المعلومات للباحثين بمقارنة تركيبة بروتين جديد مع بروتينات أخرى بطريقة أتوماتيكية و مقارنة جينات بعض الفيروسات مع فيروسات أخرى مما أمكن من تصنيف العديد من البكتيرا و الفيروسات و بالتالي إمكانية الحصول على فكرة أولية لكيفية معالجة فيروس جديد و إمكانية تحديد فصيلته.
ولضمان دقة المعلومات المخزنة في هذه القواعد أسست لجنان مختصه في تنقيح و التدقيق فيها, فمثلا إذا إكتشف فريق بحث بنية بروتين غير معروف أو تركيبة الحمض النووي لفيروس أو بكتريا أو أي كائن حي آخر يمكنها إرسال هذه المعلومة إلى قاعدة البيانات المختصة حيث يتم مراجعتها ومراجعة الطريقة التي أجريت بها التجارب ومن ثم إظافتها.
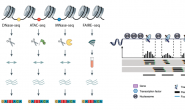
ومع تضاعف حجم هذه البيانات بطريقة أسية (exponential growth ) بدأ أداء هذه القواعد في التردي حيث أن عملية البحث أصبحت تستغرق وقتا أكبر مما إسدعي تطوير خوارزميات فعالة للمقارنة بين مختلف السلاسل.
في سنة 1970 قام نيدلمان و وينتس (Needleman–Wunsch) بتصميم خوارزمية لمقارنة سلستا بروتين ثم أتي سميث و واترمان في سنة 1981 وقاما بتطوير برنامج أسرع يعتمد على مبدا البرمجة الدينامكية.
مثال عن إيجاد أحسن مطابقة بين سلسلتين بإستعمال خوارزمية سمث و واترمان
لكن للأسف هذا لم كافيا فمع تطور تقنيات تشفير الحمض النووي و إستخلاص سلاسل البروتين أصبح بمقدور العلماء تشفير جنات تحتوي على ملايين النوكليوتيدات فأصبح من الضروري إيجاد طرق بحث فعالة أكثر للبحث عن سلسلة في وسط بحر من اللرموز. فتم تطوير برنامج بلاست (BLAST) للبحث في ظرف لا يتعدى دقائق و البحث عن سلاسل مشابهة للسلسة المرجوا البحث عنها في مختلف الكائنات. ويعتبر برنامج بلاست من البرامج الأكثر إستعمالا من طرف علماء الأحيان إلى الآن وهو يعتبر من الأدوات الأساسية لكل طالب أحياء.
مثال على نتيجت بحث بلاست
إلى غاية بدايات القرن العشريين كان إهتمام علماء المعلومات الحيوية منصبا على كيفية تحسين طرق مقارنة السلاسل والشفرات الوراثية بطرق تكون ذات معنى بيولوجي أكثر و تطوير خوارزميات لتحديد ما إذا كانت شفرة وراثية ما تمثل جينا أم لا و ما هي الأجزاء التي تكون هذا الجين,....إلخ.
رسم تمثيلي لمختلف أجزاء الجين
في سنة 1990 قام المعهد الوطني للصحة الأمريكي بإطلاق مشروع طموح يتمثل في تشفير جينوم الإنسان و وضعوا لذالك مخططا لمدة 15 سنة و ميزانية تقدر بثلاثة بلايين دولار. شارك في المشروع مئات من العلماء وبالإضافة إلى المخابر الأمريكية كان فيه عدة مخابر موزعة على 18 دولة. قد يتسآل أحد لماذا هذا العدد الهائل من الموارد البشرية و المالية؟ و الإجابة تعود لنوع تقية التشفير آن ذاك إذ كانت تعتمد على طريقة تحليل الحمض النووي المطورة من طرف فريدريك سانغر (Frederick Sanger ) والتي كانت بطيئة نحوا ما. ونظرا لكبر حجم الجينوم البشري حوالي 3.3 بليون زوج قاعدي ( يعني أحد النكليوتيدات A,C,G,T ) فقد تم تقطيع الحمض النووي إلي أجزاء تحتوي على أجزاء مشتركة و تم توزيع كل جزء على مختبر ليقوم بتشفيره بشكل مشابة للعبة تركيب الصور المقطعة.
تمكن العلماء من إكمال المشروع خمس سنوات قبل الوقت المحدد و قاموا في سنة 2000 بنشر نسخة أولية عن الحمض النووي وتم نشر النسخة النهائية سنة 2003. لكن كانت فيه في بعض الأحيان تصحيحات طفيفة و آخر تصحيح كان سنة 2009. ويمكن لأي إنسان أن يتصفح الجينوم البشري في موقع جامعة كاليفورنيا في سانتا كروز (UCSC) .
بعد نهاية المشروع ظن الناس أنها بداية النهاية و أن كل الأمراض سوف تشفر و سوف نقضي على مرض السرطان ....إلخ لكن إكتشف العلماء أنها فقط نهاية البداية و أن الطريق مازالت طويلة. حيث تم إكتشاف عدة مفاجئات, الأولي هي أن فقط نسبة واحد بالمئة من الحمض النووي تقوم بدور في الخلية أو ما يسمي الجينات أما الباقي فهو فقط نفايات مما أدى إلى ظهور عدة من التسائلات. والثاني هو أن عدد الجينة المرمزة للبرتين أقل بكثير مما كان يتوقع, حيث أن قدر العلماء في البداية وجود على الأقل 100,000 جين نظرا لتعقد بنية الإنسان لكن المفاجئة كانت أن جسم الإنسان يحتوي فقط على حوالي 20,000 إلى 25,000 جين و هو عدد أقل بكثير من عدد الجينات الموجودة في بعض الحشرات. مما أثبت أن التعقيد البنيوي للكائنات لا يعكس بالظرورة تعقيدا على مستوى الخلية.
في الجزء القادم سوف نتكلم عن كيفية تمكن العلماء من تطوير تكنولوجيات جديدة تسمح لنا الآن قرائة الشفرة الوراثية في فقط يومين وبسعر أقل بأضعاف مضاعفة من سعر مشروع الجينوم البيشري.
رابط المقالة : المعلوماتية الحيوية بالعربية » مدخل إلى علم المعلوماتية الحيوية (Bioinformatics) (الجزء الأول)