السلام عليكم,
كما راينا في المقالات السابقة, ساهمت تقنيات الجيل الجديد للسَلسلة في فتح المجال لتطوير تقنيات وطرق جديدة سمحت لنا بدراسة جوانب مختلفة من الخلية و فهم بعض الآليات البيولوجية. لكن المشكل الوحيد لهذه التقنيات هو انها تحتاج الى عدد كبير من الخلايا (آلاف او ملايين). في الكثير من الحالات يمكن الحصول على عدد كافي من الخلايا عن طريق تقنيات تنمية الخلايا, لكن هناك حالات اخرى لايمكننا فيها القيام بذلك. كما ان هناك العديد من الباحثين الذين يعتقدون ان تقنيات تنمية الخلايا قد تاثر على التعبير الجيني مما قد يؤدي الى نتائج غير دقيقة.
كما ان تقنيات السلسلة التراكمية التقليدية, كتقنية الـ Bulk RNA-seq, تعطيتنا متسوط التعبير الجيني لمجموعة من الخلايا مما قد لا يكون دقيقا لانه لا يمكن أن تتطابق خليّتان بشكل تام. خاصة اذا كانت هذه المجموعة مكونة من خليط غير متجانس من الخلايا. مثلا, ان تاخذ كل خلايا ورم سرطاني وتدرس تعبيرها الجيني كمجموعة واحدة. كما هو معروف الورم السرطاني يتكون من خلايا ذات خصائص مختلفة. فمثلا هي تحتوي على بعض الخلايا الدموية وخلايا النظام المناعي وخلايا سرطانية اخرى ذات خصائص مختلفة.

لاحظ العلماء هذا المشكل بشكل سريع, وخاصة في المجالات التي تتعامل مع عدد قليل من الخلايا كعلم الاحياء النمائي (Developmental biology) الذي يهتم دراسة المراحل الاولى للتطور الجنيني. ففي المراحل الاولى لدينا فقط بويضة ملقحة واحدة وخلال مرحلة التفلج لدينا عدد قليل من الخلايا لا يُمكننا من توفير كمية كافية من الـ RNA او DNA من اجل القيام بعملية السَلسة. وحتى وان قمنا بعملية PCR لتضخيم كميت الـ DNA او الـ RNA فربما قد نقوم بتظخيم جين على حساب اخر ونتحصل على قراءات خاطئة للتعبير الجيني.
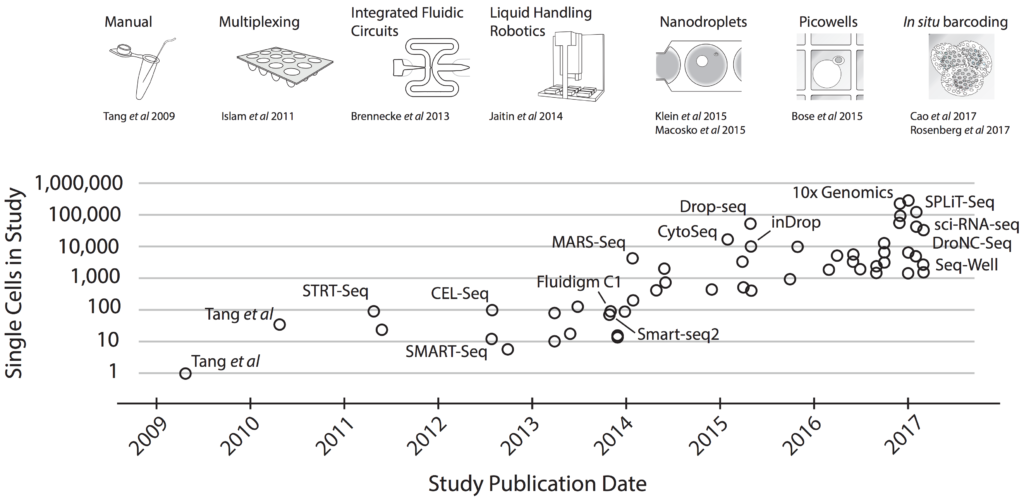
لم يدم الامر طويلا حتى بدأ الباحثون في تطوير تقنيات تسمح لنا بدراسة التعبير الجيني على مستوى الخلية الواحدة. ففي سنة 2009, ثلات سنوات فقط بعد ظهور تقنيات الجيل الجديد للسلسة, قام الباحث Tang وزملائه بتطوير طريقة تسمح بتظخيم الـ mRNA بطريقة غير متحيزة و استطاع قياس التعبير الجيني على مستوى الخلية الواحدة[1] . كما قام الباحث Navin في سنة 2011 بتطوير طريقة تسمح بسَلسلة الحمض النووي الريبوزي منقوص الأكسجين (DNA) على مستوى الخلية الواحدة [2] . هذه الاعمال والابحاث التي تلتها سمحت بفتح الباب اما مجال الـ Single-cell sequencing الذي شهد تطورا سريعا في السنوات القليلة السابقة.

مخطط يوضح بعض اهم مراحل تطور تقنية الـ Single cell sequencing (المصدر)
هذه التقنيات لم تسمح لنا فقط بدراسة التعبير الجيني او تحليل الحمض النووي على مستوى الخلايا المفردة لكن سمحت لنا ايضا بدراسة الظواهر الاجينية (epigenetic) . كما دفعت العلماء والمنظمات الى انشاء مبادرات لتحليل الخصائص البيولوجية على متسوى الخلايا المفردة كمبادرة "اطلس الخلايا البشرية" و "اطلس البيولوجية الجزئية للخلايا البشرية" و مبادرة "تشان و زوكربيرج لانشاء اطلس الخلايا البشرية"
في هذه المجموعة من المقالات سوف نحاول التعريف ببعض هذه التقنيات ومجالات استعمالاتها.
اهم مراحل الـ Single-cell sequencing
تتشابه كثيرا مراحل دراسة التعبير الجيني للخلايا المفردة مع مراحل دراسة التعبير الجيني المتبعة في الطرق التقليدية كـ RNA-seq كما هو موضح في المخطط ادناه. لكن الاختلاف الكبير يكمن في كيفية القيام بهذه المراحل. في هذه الفقرة سوف نقوم بالتعريف بأهم التقنيات المستعملة.

مخطط يوضح اهم مراحل القيام بالسلسلة على مستوى الخلية المفردة
أ. مرحلة عزل الخلايا
تعتبر اهم مرحلة من مراحل دراسة الخلايا المُفردة. تهدف هذه المرحلة الى عزل كل خلية لحالها بحيث ان نتمكن من استخراج الـ DNA او الـ RNA دون ان يمتزج مع DNA او RNA الخلايا الاخرى ودون ان نؤدي الى تغير في حالة الخلية. من الاهداف المهمة ايضا هو العزل المتجانس لكل انواع الخلايا المتواجدة في النسيج الخلوي تحت الدراسة. حيث اننا لا نعزل انواع الخلايا المتواجدة بكثرة فقط, بل نتمكن من عزل الخلايا النادرة التي تتمثل نسبة قليلة من النسيج الخلوي (مثلا 1%).
طورت عدة طرق للقيام بهذه العملية. منها ماهو يدوي ومنها ماهو اوتوماتيكي ومنها ماهو مزيج بين يدوي واتوماتيكي. بصفة عامة يمكننا تصنيف هذه التقنيات الى المجموعات التالية:
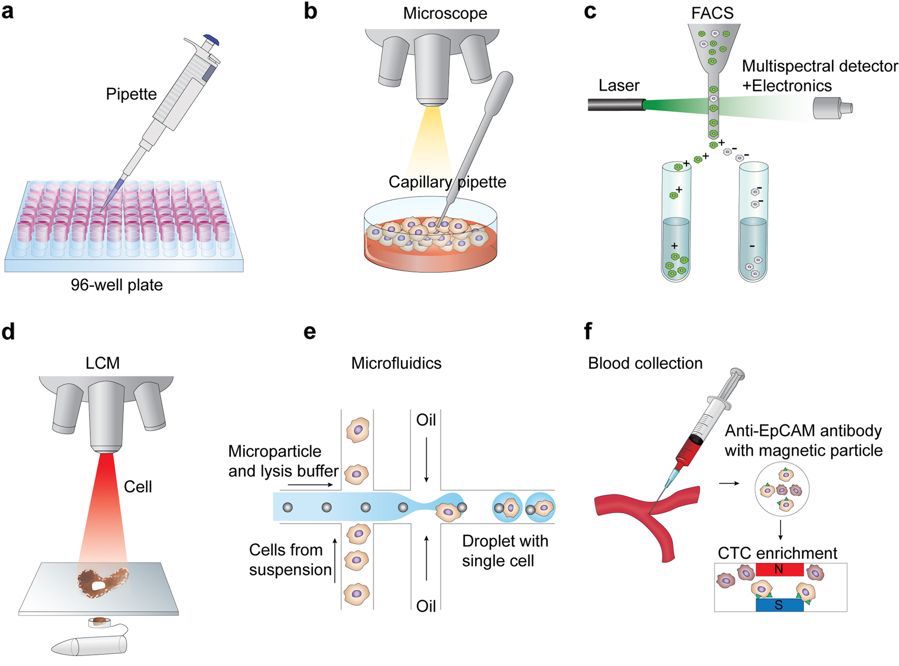
- تقنيات العزل اليدوي باستعمال الماصة:
تعتبر من التقنيات السهلة و الغير مكلفة, حيث يتم خلالها وضع كمية قليلة من الخلايا في كمية كبيرة من المحلول بحيث تكون الخلايا متشتة وبعيدة عن بعضها البعض. بعدها يتم استعمال الماصة (Pipette) ووضع جزء من المحلول في كل ثقب من صفيحة المعايرة الدقيقة (microwell plates) بحيث يكون لدينا خلية في كل ثقب موضح في الصورة a اعلاه . كما يمكننا ايضا استعمال الماصات الشعرية من اجل التقاط الخلايا واحدة بواحدة (كما هو موضح في الصورة b)
- تقنيات العزل عن طريق فرز الخلايا المستحثة بواسطة الوميض الفلورسيني (Fluorescence-activated cell sorting)
تعتبر طريقة فرز الخلايا المستحثة بواسطة الوميض الفلورسيني (FACS) من بين الطرق الشائعة نظرا لتوفر جهاز قياس التدفق الخلوي في الكثير من المختبرات البيولوجية مما يجعله من بين الطرق المتاحة و الغير المكلفة نسبيا. في هذه الطريقة يتم استعمال مضاد حيوي مشع لتعليم الخلايا التي تحتوي على بروتين معروف مسبقا من اجل فرز خليط غير متجانس من الخلايا البيولوجية الى مجموعتين : مجموعة الخلايا التي تحتوي على البروتين المعروف والخلايا التي لاتحتوي على هذا البروتين (كما هو موضح في الصورة c).
من بين سلبيات هذه التقنية هو حاجتنا الى استعمال عدد كبير من الخلايا (على الاقل عشرة آلاف خلية) والذي لايمكن القيام به لكل انواع الخلايا. ايضا تحتاج الى استعمال مضاد حيوي والذي لايمكن توفيره او معرفته لكل انواع الخلايا.
- تقنيات العزل باستعمال ملتقط الليزر (Laser capture microdissection)
تعتمد هذه التقنية على استعمال ملتقط الليزر من اجل عزل الخلايا بدقة من الانسجة الصلبة دون إتلافها (كما هو مبين في الصورة d). تتميز هذه التقنية بغلائها لكنها تحفظ المعلومات حول وضعية الخلية داخل النسيج مما يسمح لنا من دراسة التعبير الجيني والموقع الفيزيائي للخلايا والعلاقة بينها.
- تقنيات العزل باستعمال علم الموائع الدقيقة (Microfluidics)
تعتبر الآت عزل الخلايا المفردة باستعمال علم الموائع الدقيقة (Microfuidics) من بين التقنيات الاكثر شهرة واستعمالا في مجال دراسة الخلاية المفردة. ساهم ظهور هذه التقنايات الى ثورة في مجال الـ Single-cell sequencing, نظرا لتكلفتها الرخيصة نسبيا وقدرتها على عزل مئات حتى آلاف الخلايا في تجربة واحدة. لم تلاقي هذه التقنيات القبول في المجال الاكاديمي فقط, بل بدات بغزو المجال الالينيكي والتجاري.
تنقسم التقنيات المبنية على اساس علم الموائع الدقيقة بدورها الى عدة اقسام لن نتطرق اليها في هذا المقال من اراد تعريف شامل بهذه التقنيات يمكنه الاطلاع على المصدر رقم 5 في اسفل الصفحة.
تعتبر التقنيات التي تعتمد على القطيرات من بين التقنيات الاكثر رواجا, حيث انها تعتمد على عزل كل خلية في قطيرة واحدة تحتوي على كرية تظخيم (bead) ومجموعة من الانزيمات تساعد على تحليل الخليلة وتظخيم الـ RNA او الـ DNA في كل خلية لحالها (كما هو موضح اسفله).

- والعديد من التقنيات الاخرى
على الرغم من مرور حوالي عشر سنوات على ظهور هذا المجال, الا انه لا زال يشهد تطور سريعا من الجانب التقني وجانب تحليل البيانات. فمن الجانب التقني يريد الباحثون تطورير تقنيات جديدة تسمح لنا بدراسة جوانب اخرى من الخلية, كالتغيرات الاجينية و تعبير البروتينات بطريقة اكثر دقة اواقل تكلفة.
ب. مرحلة التظخيم وتحضير المكتبات
هذه المرحلة تعدف الى توليد مجموعة سلاسل من اجل السَلسَة والحصول على البيانات. من بين الخطوات الازمة في هذه المرحلة هي استعمال انزيمات من اجل تحليل الخلية (cell lysis), ثم القيام بعملية النسخ العكسي وانشاء cDNA, تهجين السلسلة المعاكسة والقيام بعملية التظخيم.
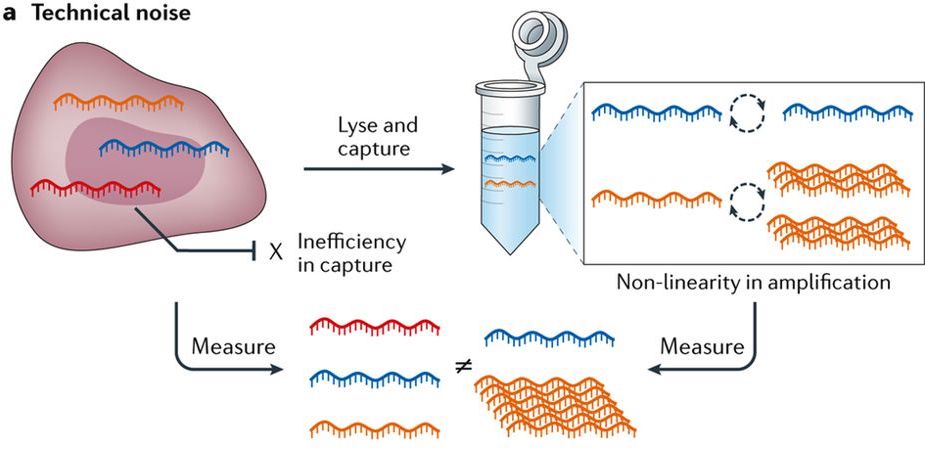
لو اخذنا تقنيات الـ Microfluidics كمثال (كما هو مبين اسفله), تعتمد تقنيات الـ Single-cell sequencing على عزل كل خلية مع مجموعة من الانزيمات التي تقوم بتحليها بحيث تسمح بتحرير الـ mRNA. تلتصق الذيول عديدة الاديلينات (Poly(A) tail) لهذه الـ mRNA بكرية (bead) يحتوي على مجموعة من البوادي. هذه البوادي تحتوي على باركود (cell barcode) وحيد لكل خلية يسمح لنا بمعرفة الخلية التي اتت منها هذا السلاسل و باركود وحيد (unique molecular identifiers او UMIs) لكل mRNA يمكننا من معرفة الـ mRNA الذي اتت منه السلسة.

قل يتسائل البعض عن اهمية اضافة الـ UMI مادام لدينا باركود يمكننا من معرفة الخلية التي اتت منها السلاسل. في الحقيقة, تم اظافته لتفادي مشاكل التظخيم الناتجة عن استعمال PCR حيث انه لايتم تضخيم كل الجينات بطريقة مشابهة. حيث انها بعضها يتم تضخيمه اكثر من الآخرين (كما هو مبين اسفله). بالاضافة الى هذا المشكل هنا مشاكل تقنية اخرى تتمثل في ان تقنيات الـ Single-cell sequencing تسمح لنا فقط بالالتقاط 10% ~ 20% من الترنسكريبتوم حيث انه لايمكن ان تلتصق كل الـ mRNA بالكرية خاصة الـ mRNA ذات العدد القليل من النسخ في الخلية.
ج. السَلسلة
هذه المرحلة لا تختلف عن تقنية الـ RNA-seq حيث يتم السلسلة بطريقة عادية للمكتبات. الفرق الوحيد في بعض الاحيان هو طول السلاسل المتحصل عليها حيث تختلف من تقنية الى اخرى ويجب اخبار التقني المسؤول عن السلسلة بذلك.
د. تحليل البيانات
تعتبر هذه المرحلة من اهم المراحل وفي بعض الاحيان تكون معقدة قليلا. لكي لا نطيل كثيرا في هذا المقال, سوف نتطرق الى هذه المرحلة في المقالات القادمة.

بعض المصادر
- mRNA-Seq whole-transcriptome analysis of a single cell. Tang F et al. Nat Methods. 2009 May; 6(5):377-82.
- Tumour evolution inferred by single-cell sequencing. Navin N et al. Nature. 2011 Apr 7; 472(7341):90-4.
- https://arabicedition.nature.com/journal/2017/08/547125a/تبسيط-التسلسل-الجيني-للخلايا-المُفْرَدَة
- Advances and Applications of Single Cell Sequencing Technologies.Yong Wang and Nicholas E. Navin. Mol Cell. 2015 May 21; 58(4): 598–609.
- Scaling by shrinking: empowering single-cell 'omics' with microfluidic devices. Sanjay M. Prakadan, Alex K. Shalek & David A. Weitz. Nature Reviews Genetics.2017.
رابط المقالة : المعلوماتية الحيوية بالعربية » مدخل الى مجال تحليل الخلايا المفردة Sing-cell sequencing (الجزء الاول)