بسم الله الرحمن الرحيم
لقد تمكنا من خلال المقال السابق من فهم مبدأ عمل الخلية بصفة عامة ورأينا أن نشاط كل جين يؤدي إلى إنتاج بروتين والذي يقوم بعدة أدوار في الخلية. هذا المبدأ يسمى في علم الأحياء الجزيئ بالمبدا الرئيسي (Central Dogma) الذي يوصف في العادة ب:
من هنا يمكننا أن نستنتج أنه كلما زاد نشاط جين من الجينات زادت أولا كمية الـ mRNA (الـ RNA الرسول) المنتجة وبالتالي تزيد نسبة البروتينات المصنعة من طرف هذا الجين. يعني أنه إذا أردنا أن نتعرف على الجينات النشطة في الخلية يمككننا أن نأخذ كل البروتينات في الخلية ونرى من هو البروتين الموجود بأكبر نسبة. هذه الطريقة تبدو منطقية لكن من الجانب العملي تمثل عدة تحديات, فأولا بنية البروتين مختلفة عن بنية الجين وتحليلها لايزال صعبا وبالتالي فإن تحليل آلاف البروتينات سوف يأخذ وقتا كبيرا كما أننا أيضا رأينا أن الحمض الأميني الواحد يمكن تشفيره من طرف عدة كودونات, يعني حتى لو عرفنا تسلسل الأحماض الأمينية في البروتين سوف يكون لدينا عدة إحتمالات لصيغة الجين الذي أنتج هذا البروتين.
الطريقة الأسهل هي إستخلاص ال mRNA الموجود في الخلية وقياس نسبته , إذا أن بنيته مكملة لبنية الجين وله خاصية هامة وهو أنه قابل للتهجين (Hybridization) , يعني أنه يمكننا أن نصنع نسخة مكملة له (cDNA) بإستعمال الجزيئات القاعدية C,G,T,A.
تقنية ال Micrro array تعتمد على هذا المبدأ (مبدأ التهجين) من أجل قياس نسبة نشاط الجينات بطريقة متوازية بحيث تمكننا في تجربة واحدة من قياس نسبة نشاط كل الجينات الموجودة في الجينوم البشري بسعر بسيط. فتقنية الـ Microarray تقول لك حسنا, بما أننا سوف نقوم بقياس نسبة الـ mRNA فما علي إلا أن أحضِّر شريحة وأضع فيها سلاسل مكملة لسلاسل الـ mRNA التي يمكن للخلية إنتاجها وبالتالي عندما أضيف الـ mRNA إلى الشريحة فسوف تلتصق بالسلسلة التي تكملها, ولمعرفة كم نسخة تم إنتاجها من كل جين أقوم بوضع نسخ كثيرة من كل cDNA وأقيس نسبة الـ mRNA التي إلتصقت (هجن) مع السلاسل المكملة. ولقياس هذه النسبة أقوم بإضافة عنصر مشع لكل mRNA وبعدها أقيس في الشريحة نسبة إضائة كل نقطة.
قد يتسآل البعض : وكيف يمكن معرفة سلسلة الـ cDNA قبل إستخلاص الـ mRNA ؟ في تقنية الرقائق الدقيقة نقوم فقط بدراسة الجينات التي نعرف تسلسلها و الآن مع تشفير جينوم عدة كائنات لدينا اللآن مجموعة من قواعد البيانات التي تحتوي على صيغ السلاسل المكملة والتي يمكن تحميلها بسهولة من النت.
رسم بياني يوضح مبدأ التهجين, حيث يرتبط كل mRNA بالسلسلة المكملة cDNA (المصدر)
تم تطوير هذه التقنية في نهاية التسعينات ولاقت رواجا كبيرا إلى حد اللآن رغم ظهور تقنيات جديدة إلا أن الـ Microarray لاتزال تمثل تقنية كلاسيكية في كل المختبرات (على الأقل في الدول المتطورة).
توجد عدة طرق من أجل صناعة هذه الشرائح, الطريقة الأولى هي إستعمال روبوتات (Spotting robot) تقوم بتثبيت السلاسل المكملة في الفتحات (المسابير أو Probs) الموجودة في الشريحة. وفي هذه الطريقة يتم أولا تجهيز مكتبة السلاسل التكميلية (cDNA library) ووضعها في الروبوت وإخباره بمكان وضع كل سلسلة.
صورة عن روبوت (Spotting robot) يقوم بتثبيت السلاسل المكملة على الشريحة (المصدر)
الطريقة الثانية تتمثل في بناء السلاسل التكميلية جزئ قاعدي بجزء قاعدي مباشرة على الشريحة (in-sito Spotting) , حيث يضاف في كل مرة جزء قاعدي إلى السلاسل بشكل متوازي. كما هو موضح في الصورة, يتم إستعمال الشريحة و قناع. الشريحة في وضعها الأولي تحتوي على سلاسل مكونة من مجموعة هيدروكسيل وجزء حامي يمنع إلتصاق مركبات أخرى. و في كل مرة يتم إستعمال قناع يحتوي على فراغات تحدد المكان الذي نريد إضافة الجزيئ القاعدي إليه, مثلا T, ثم يقوم تحفيز الأماكن المحددة بإستعمال الضوء مما يؤدي إلى نزع الجزء الحامي (ملون بالأحمر في الصورة) وبعدها بتم وضع المركب T على الشريحة فيلتصق فقط في الأماكن المفتوحة, بعدها يتم غسل الشريحة لنزع المكونات التي لم تلتصق ويغير القناع و تضاف قواعد أخرى.
رسم بياني يوضح مبدأ تقنية in-Sito Spotting (المصدر)
تستخدم هذه التقنية في الشرائح المطورة من طرف شركة .Affymetrix Inc وهي الشريحة الأكثر إستعمالا, إذ تسمح بتحكم أكبر بالسلاسل التي يراد إضافاتها.
صورة عن شريحة مصنعة من طرف Affymetrix Inc (المصدر)
كيف يتم إستعمال رقائق الحمض النووي من أجل دراسة الأمراض ؟
كما ذكرنا سابقا, أن هذه الرقائق تستعمل من أجل قياس نسبة نشاط الجين ومعرفة الجينات النشطة و الخامدة. لكن عندما تكون الخلية في حالة غير طبيعية ( السرطان مثلا) فإن نشاط الجينات سوف يتغير, فبعض الجينات التي يجب إن تكون نشطة يقل نشاطها بصفة ملحوظة أو ينعدم تقريبا أو من الجهة الأخرى يتم تنشيط جينات التي يجب أن تكون ذات نشاط ضعيف أو غير نشطة.
في العادة من أجل دراسة هذا التغيير نقوم بالخطوات التالية:
- تحضير العينات:
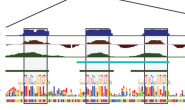
في هذه المرحلة يتم أخذ عينات من خلايا لأناس أصحاء (مثلا خلايا الكبد) و عينات من خلايا أناس مرضى (بطبيعة الحال تكون من نفس النوع, في هذه الحالة خلايا كبد مصابة). في العادة يتم أخذ عينات من عدة مرضي وأناس أصحاء لتفادي الأخطاء الناتجة عن العملية وعن نوعية الخلايا.
- إستخراج الـ mRNA:
بعد الحصول على العينات يتم إستخراج الـ mRNA من خلايا كل عينة ووضع mRNA العينات العادية في أنبوب و mRNA العينات المريضة في أنبوب آخر. في بعض الحالات يتم تحويل الـ mRNA إلى cDNA وتكون السلسلة الموجودة في الشريحة هي السلسلة الحقيقية للجين, لكن كلها تتبع نفس المبدأ. في هذا المقال سوف نستعمل الـ mRNA.
- توسيم الـ mRNA:
في هذه المرحلة يتم توسيم الـ mRNA باستعمال مكونات كيميائة تشع عندما تحفز بإستعمال الليزر, فيتم توسيم واحد من الأنابيب بالمكون Cy3 ( الذي يشع عند إستعمال الليزر الأخضر) و الأنبوب الآخر بالمكون Cy5 (الذي يشع عند إستعمال الليزر الأحمر). ثم بعدها نقوم بوضع محتوى الأنبوبين معا في أنبوب واحد.
- التهجين:
بعد إضافة الموسمات نقوم بإضافة الـ mRNA الموسمة إلى الـ Micrroarray, للتم بعدها عملية التهجين بحيث تلتصق كل mRNA بالسلسلة المكملة لها وفقا لمبدأ التهجين الذي شرحناه سابقا.
- غسل الرقاقة:
يتم بعدها غسل الرقاقة من أجل الـ mRNA التي لم تلتحم مع مكملاتها من أجل تفادي أية أخطاء في قياس نسبة النشاط.
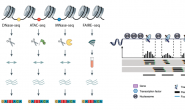
- المسح بالليزر وتحليل الصور:
كما قلنا سابقا فإن الـ mRNA تكون موسومة بلونين مختلفين (ليكن الأخضر للخلايا العادية و الأحمر للخلايا المريضة) حيث يشع كل لون عند إستعمال ليزر معين. فلقياس نسبة الجينات في الخلايا العادية نقوم بإرسال الليزر الأخضر فتشع الجينات العادية, فنأخذ صورة على هذا الإشعاع, ثم نقوم بإستعمال الليزر الأحمر فتشع الجينات النشطة في الخلايا المريضة. نقوم بعدها بأخذ هذه الصور ومعالجتها رقميا لمعرفة نسبة إشعاع كل جين في كل من العينتين. ولضمان أن القياسات المتحصل علها دقيقة يجب ضمان أن كل نقطة في من الرقيقة تتعرض لنفس نسبة الضوء وأن الصورة مأخوذة بشكل واضح.
في العادة يتم دمج الصورتين للحصول على فكرة أولية عن الجنينات النشطة والغير نشطة في الخلايا المريضة. فمثلا بعد دمج الصورتين ولآحظنا أن لون أحد المسابير مائل للون الأخضر فهذا يعني أن نشاط الجين في الخلية العادية أكثر من نشاطه في الخلية المريضة وإذا رأينا أن مسبارا له لون أحمر فنستنتج أن هذا الجين أكثر نشاطا في الخلايا المريضة أما إذا كان لون المسبار أصفر فنستنتج أن نشاط هذا الجين لم يتغير.
لكن بطبيعة الحال لايمكن الحكم فقط بإستعمال العين المجردة بل يجب القيام ببعض التحاليل الإحصائية لكي نتأكد أن هذا التغير كان تغيرا ذا معنى وماهو إحتمال ألا يكون ناجما عن الصدفة. وبما أن التجارب تكون في محيطات مختلفة فربما كان هناك بعض الإختلافات الناجمة عن كيفية أداء التجربة وليس عن نشاط الجين. لهذه الأسباب وغيرها توجب علينا تطوير بعض الطرق الإحصائية و الحسابية من أجل التأكد أن النتائج المتحصل عليها مقاربة للصحة.
مخطط يشرح مراحل إستعمال الـ Microarray من أجل المقارنة بين نشاط الجينات بين عينتتن (المصدر)
صورة تبين بعض المشاكل التي يمكن أن تحدث عند أخذ الصور (مأخوذة من درس البروفيسور Xuegong, Zhang)
مثال عن معالجة المعلومات عن نشاط الجينات والعلاقة بينهم (المصدر)
بطبيعة الحال الحلم الأكبر أن تصبح هذه التقنيات جزءا من العلاج الروتيني في المستشفيات حيث يقوم الطبيب بالحصول على تقرير على المستوى الجيني للمريض فيمكنه بذالك معرفة كمية ونوع الدواء الذي يناسب المريض وهو مهم جدا خاصة لمرضى السرطان حيث يمكن تحديد كمية العلاج الكيمياي المناسب والذي يسبب أقل أضرار جانبية,...إلخ. بإختصار الدخول في عصر الطب الشخصي (Personalized medicine) و العلاج الجيني (Gene Therapy) . لهذا يتطلب تطوير طرق حسابية دقيقة وموثوقة من أجل تجنب أية أخطاء.
في الجزء القادم سوف أحاول شرح بعض الطرق الإحصائية لتصحيح القياسات ومعالجة البيانات. أتمنى أن يكون هذا المقال مفيدا وسهلا للفهم.
مصادر:
- DOV STEKEL, Microarray Bioinformatics, Cambridge University Press, 2003
- Christine A.W. and Lois A.S , A guide to issues in microarray analysis: application to endometrial biology, Reproduction 130 (1) 1-13
- G. Hardiman, Microarray platforms—Comparisons and contrasts, Pharmacogenomics, 5 (5) (2004), pp. 487–502
- Adi L. et al, Analysis of microarray experiments of gene expression profiling, Am J Obstet Gynecol. Aug 2006; 195(2): 373–388
- http://www.affymetrix.com/
رابط المقالة : المعلوماتية الحيوية بالعربية » تقنية رقائق الحمض النووي الدقيقة (DNA Microarray) {الجزء الأول}